BJN#126 – Epitope Matching : vers l’avenir du HLA en transplantation ?
Class II HLA Epitope Matching—A Strategy to Minimize De Novo Donor-Specific Antibody Development and Improve Outcomes
Merci à Christophe Masset, interne en néphrologie à Nantes et membre du Comité Scientifique du CJN, pour cette synthèse bibliographique. Vous aussi, n’hésitez pas à nous envoyer vos lectures !
Introduction
Le rejet humoral diminue de 40% à 10 ans la survie greffon, particulièrement quand il est lié aux DSA de classe II. La survenue de DSA de novo (dnDSA) est liée au mismatching HLA donneur-receveur, actuellement basée sur les antigènes ou allèles HLA. L’évaluation des mismatchs épitopiques permet une approche plus immunologique, dans laquelle tous les sites immunogènes de la molécule HLA sont évalués. Cette étude de cohorte compare les mismatchs HLA “classiques” et épitopiques de patients ayant développé des DSA de classe II.
Patients/matériels et méthodes
286 patients sans DSA prégreffe, transplantés à Winnipeg (Canada) entre 1999 et 2008 ont eu un monitoring de DSA post greffe (MFI ≥ 300, évalué par la méthode Single Antigen). Le nombre de Mismatchs HLA donneur-receveur allélique (HLA-DR évalué à l’aide d’un score HLA-DRβ1/3/4/5 allant de 0 à 4 mismatchs ; et de même manière un score HLA-DQα1/β1 de 0 à 4 mismatchs) et le nombre de Mismatchs épitopique* (défini à l’aide de HLAMatchmaker) des patients ayant développé des dnDSA était comparé à ceux des patients n’ayant pas développé de dnDSA. Des tests de régression logistique étaient réalisés afin de définir les facteurs associés avec la survenue de dnDSA.
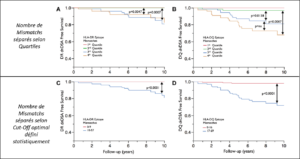
Résultats
16% des patients (45/286) ont développé un dnDSA de classe II (MFI médiane = 5266) : 9 anti-DR, 24 anti-DQ, 12 anti-DR et anti-DQ, aucun anti-DP. Il y avait une tendance non significative à un plus grand nombre de mismatchs alléliques pour les patients ayant développé un anti-DR ou un anti-DQ par rapport aux patients n’ayant pas développé de dnDSA. En comparaison, le nombre de mismatchs épitopiques était significativement plus élevé (HLA-DR : 21,4 vs. 13,2, p<0,02 ; HLA-DQ : 27,5 vs. 17,3, p<0,001). Pour les patients qui ont développé des anti-DR et anti-DQ, les mismatchs alléliques étaient significativement plus élevés (HLA-DR : 2,6 vs. 1,8, p<0,02 ; HLA-DQ : 2,7 vs. 1,9, p<0,05), de même que les mismatchs épitopiques (HLA-DR (24,2 vs. 13,2, p<0,01) and HLA-DQ (28,2 vs. 17,3, p<0,01).
L’effet des mismatchs épitopiques sur la survie sans dnDSA est représenté sur les courbes Kaplan Meier de la figure jointe (séparation du nombre mismatchs par quartiles sur les figures A et B, et selon un cut-off optimal défini statistiquement sur les figures C et D).
Conclusion
Le Matching épitopique HLA parait plus efficace que le Matching antigénique actuellement utilisé pour prédire – et donc éviter – le développement de dnDSA anti-Classe II. L’utilisation du système épitopique en lieu et place du système antigénique actuellement en place est plus proche du fonctionnement réel du système immunitaire, et devrait être utilisé pour l’évaluation du risque immunologique par les équipes de transplantation dans les prochaines années.
* Le système immunitaire développe des anticorps après reconnaissance d’antigènes « non-soi », et particulièrement lors de la reconnaissance d’épitopes présents sur ces antigènes. L’affinité de fixation épitopique est déterminée par un petit nombre d’Acides Aminés, présents au centre de l’épitope. HLAMatchmaker compare les séquences polymorphiques d’Acide Aminés entre les allèles donneur et receveur pour identifier et quantifier les différences. Seuls les séquences polymorphiques d’AA d’interet et les AA présents à la surface et donc accessibles à la fixation antigénique (ou à proximité de la surface) sont considérés. HLAMatchmaker utilise une représentation 3D de la localisation des AA sur la structure de la molécule HLA afin d’identifier les séquences d’AA exposées à la surface, mais aussi d’éventuelles patchs polymorphiques d’AA qui seraient discontinus dans la séquence linéaire mais proches géographiquement au niveau de la structure 3D. Les patchs polymorphiques d’AA à la surface sont appelés eplets. A l’aide de cette méthode, un catalogue des eplets (dérivés de la fixation épitopique) est établi pour chaque locus HLA.
Les plus du papier
- Une des premières études à montrer l’importance clinique du système HLA Epitopique
- Un raisonnement immunologique beaucoup plus proche de la réalité
Les critiques
- Peu de patients
- L’ensemble des éplets des antigènes HLA n’est pas encore définie et incluse dans HLAMatchmaker

